R入門
カルマンフィルタ(共立出版)
最終更新:
r-intro
目次
体重の計測値に関するフィルタ化推定量とその95%信頼区間(p.29)
公式ページからダウンロードした入力ファイル「Weight.dat」をカレントディレクトリに置いておくこと。
> dtf <- read.table("Weight.dat", header = FALSE)
> yt <- dtf$V1
> n <- nrow(dtf)
> tn <- 1:n
> #
> mod <- SSModel(yt ~ SSMtrend(1, Q = NA), H = NA)
> fit <- fitSSM(mod, numeric(2), method = "BFGS")
> kfs <- KFS(fit$model)
> at <- kfs$a[c(-1)]
> ppt <- kfs$P[, , -1] - as.vector(fit$model$Q)
> alp <- 0.05
> atcon1 <- at + qnorm(alp / 2) * sqrt(ppt)
> atcon2 <- at + qnorm(1 - alp / 2) * sqrt(ppt)
> #
> png("kalmanfil_p029.png", width = 768, height = 512)
> plot(tn, yt, type = "n", ylim = c(83, 87), xlab = "経過日数", ylab = "体重(kg)")
> lines(tn, atcon1, lty = "solid", lwd = 1)
> lines(tn, atcon2, lty = "solid", lwd = 1)
> lines(tn, at, lty = "solid", lwd = 2)
> lines(tn, yt, lty = "dotted")
> points(tn, yt, pch = 21, bg = "white", cex = 1.3)
> dev.off()
null device
1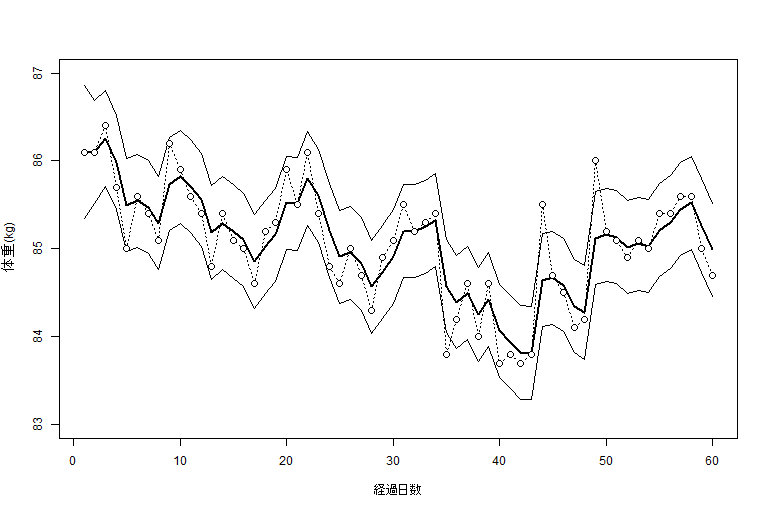
ローカルレベルモデルによる体重の計測値に関する平滑化状態とその95%信頼区間(p.33)
公式ページからダウンロードした入力ファイル「Weight.dat」をカレントディレクトリに置いておくこと。
> dtf <- read.table("Weight.dat", header = FALSE)
> yt <- dtf$V1
> n <- nrow(dtf)
> tn <- 1:n
> #
> mod <- SSModel(yt ~ SSMtrend(1, Q = NA), H = NA)
> fit <- fitSSM(mod, numeric(2), method = "BFGS")
> kfs <- KFS(fit$model)
> aht <- kfs$alphahat
> vvt <- as.vector(kfs$V)
> alp <- 0.05
> ahtcon1 <- aht + qnorm(alp / 2) * sqrt(vvt)
> ahtcon2 <- aht + qnorm(1 - alp / 2) * sqrt(vvt)
> #
> png("kalmanfil_p033.png", width = 768, height = 512)
> plot(tn, yt, type = "n", ylim = c(83, 87), xlab = "経過日数", ylab = "体重(kg)")
> lines(tn, ahtcon1, lty = "solid", lwd = 1)
> lines(tn, ahtcon2, lty = "solid", lwd = 1)
> lines(tn, aht, lty = "solid", lwd = 2)
> lines(tn, yt, lty = "dotted")
> points(tn, yt, pch = 21, bg = "white", cex = 1.3)
> dev.off()
null device
1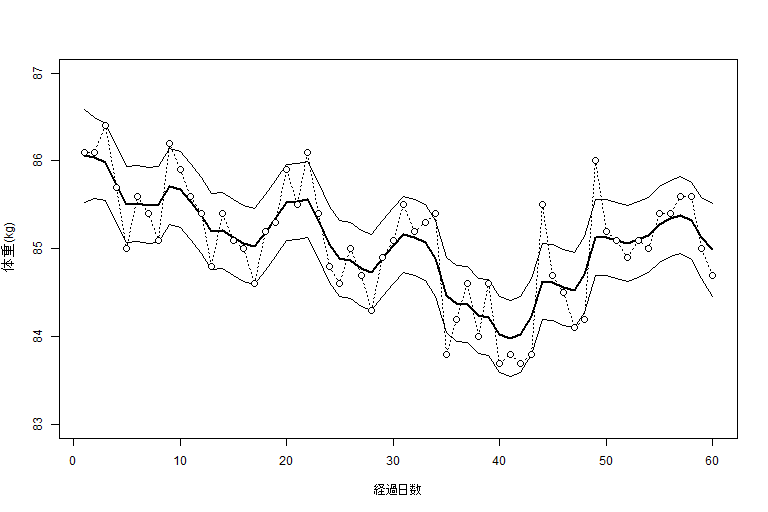
ローカルレベルモデルによる体重の計測値(欠測含む)に関する平滑化状態とその95%信頼区間と95%予測区間(p.37)
公式ページからダウンロードした入力ファイル「Weight.dat」をカレントディレクトリに置いておくこと。細実線が95%信頼区間。破線が95%予測区間。
> library(KFAS)
> dtf <- read.table("Weight.dat", header = FALSE)
> yt <- yt0 <- dtf$V1
> n <- nrow(dtf)
> t <- 1:n
> mv <- 21:40
> yt[mv] <- NA
> mod <- SSModel(yt ~ SSMtrend(1, Q = NA), H = NA)
> fit <- fitSSM(mod, numeric(2), method = "BFGS")
> kfs <- KFS(fit$model)
> aht <- kfs$alphahat
> vvt <- as.vector(kfs$V)
> seps2 <- as.vector(fit$model$H)
> alp <- 0.05
> ahtcon1 <- aht + qnorm(alp / 2) * sqrt(vvt)
> ahtcon2 <- aht + qnorm(1 - alp / 2) * sqrt(vvt)
> ahtpre1 <- aht + qnorm(alp / 2) * sqrt(vvt + seps2)
> ahtpre2 <- aht + qnorm(1 - alp / 2) * sqrt(vvt + seps2)
> plot(t, yt, type = "n", xlab = "経過日数", ylab = "体重(kg)")
> lines(t, yt0, lty = "dotted", col = "gray")
> points(t, yt0, pch = 21, bg = "white", col = "gray", cex = 1.3)
> lines(t[mv], ahtpre1[mv], lty = "dashed", lwd = 3)
> lines(t[mv], ahtpre2[mv], lty = "dashed", lwd = 3)
> lines(t, ahtcon1, lty = "solid", lwd = 1)
> lines(t, ahtcon2, lty = "solid", lwd = 1)
> lines(t, aht, lty = "solid", lwd = 2)
> lines(t, yt, lty = "dotted")
> points(t, yt, pch = 21, bg = "white", cex = 1.3)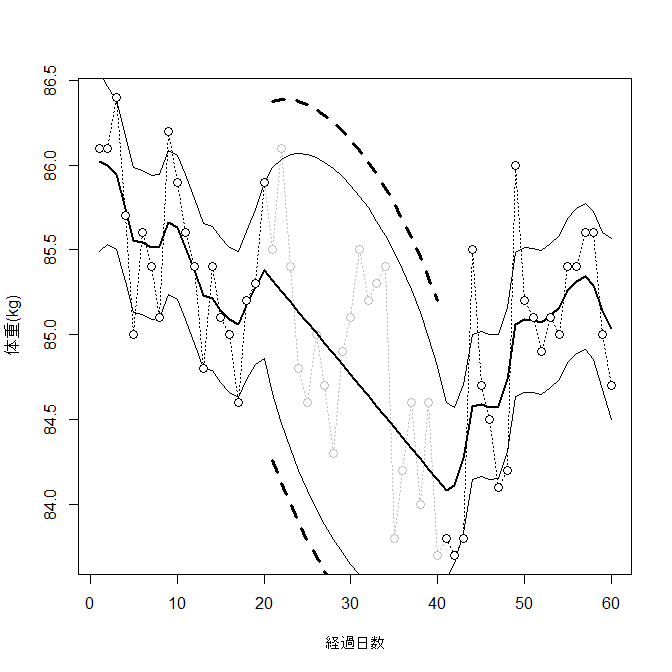
トレンドのあるモデルの比較(水準成分と傾き成分の推移)(pp.76-79)
公式ページからダウンロードした入力ファイル「Weight.dat」をカレントディレクトリに置いておくこと。
> library(KFAS)
> dtf <- read.table("Weight.dat", header = FALSE)
> yt <- dtf$V1
> # ローカルレベルモデル 灰実線
> mod1tr <- SSModel(yt ~ SSMtrend(1, Q = NA), H = NA)
> fit1tr <- fitSSM(mod1tr, numeric(2), method = "BFGS")
> kfs1tr <- KFS(fit1tr$model)
> aht1tr <- kfs1tr$alphahat[, "level"]
> # 2次のトレンドモデル 黒実線
> mod2tr <- SSModel(yt ~ SSMtrend(2, Q = c(list(0), list(NA))), H = NA)
> fit2tr <- fitSSM(mod2tr, numeric(2), method = "BFGS")
> kfs2tr <- KFS(fit2tr$model)
> aht2tr <- kfs2tr$alphahat[, "level"]
> ahts2tr <- kfs2tr$alphahat[, "slope"]
> # ローカル線形トレンドモデル 黒破線
> modllt <- SSModel(yt ~ SSMtrend(2, Q = c(list(NA), list(NA))), H = NA)
> fitllt <- fitSSM(modllt, numeric(3), method = "BFGS")
> kfsllt <- KFS(fitllt$model)
> ahtllt <- kfsllt$alphahat[, "level"]
> ahtsllt <- kfsllt$alphahat[, "slope"]
> #
> png("kalmanfil_fig3_1a.png", width = 768, height = 512)
> plot(yt, lty = 3, type = "o", xlab = "経過日数", ylab = "水準成分")
> lines(aht1tr, lwd = 2, col = 8) # ローカルレベルモデル(灰実線)
> lines(aht2tr, lwd = 2) # 2次のトレンドモデル(黒実線)
> lines(ahtllt, lwd = 2, lty = 2) # ローカル線形トレンドモデル(黒破線)
> dev.off()
null device
1
> #
> png("kalmanfil_fig3_1b.png", width = 768, height = 512)
> plot(ahts2tr, lwd = 2, xlab = "経過日数", ylab = "傾き成分") # 2次のトレンドモデル(黒実線)
> lines(ahtsllt, lwd = 2, lty = 2) # ローカル線形トレンドモデル(黒破線)
> dev.off()
null device
1
> #
> r <- qv <- seta12 <- seta22 <- seps2 <- mll <- aic <- mse <- double(3)
> r <- c(2, 2, 3)
> qv <- c(1, 2, 2)
> seta12[1] <- as.vector(fit1tr$model$Q[, , 1])
> seta12[2] <- as.vector(diag(fit2tr$model$Q[, , 1])[1])
> seta22[2] <- as.vector(diag(fit2tr$model$Q[, , 1])[2])
> seta12[3] <- as.vector(diag(fitllt$model$Q[, , 1])[1])
> seta22[3] <- as.vector(diag(fitllt$model$Q[, , 1])[2])
> seps2[1] <- as.vector(fit1tr$model$H[, , 1])
> seps2[2] <- as.vector(fit2tr$model$H[, , 1])
> seps2[3] <- as.vector(fitllt$model$H[, , 1])
> mll[1] <- kfs1tr$logLik - sum(kfs1tr$Finf > 0) * log(2 * pi) / 2
> mll[2] <- kfs2tr$logLik - sum(kfs2tr$Finf > 0) * log(2 * pi) / 2
> mll[3] <- kfsllt$logLik - sum(kfsllt$Finf > 0) * log(2 * pi) / 2
> aic <- -2 * mll + 2 * (r + qv)
> idx <- 3:60
> n <- length(idx)
> mse[1] <- sum(kfs1tr$v[idx] ^ 2) / n
> mse[2] <- sum(kfs2tr$v[idx] ^ 2) / n
> mse[3] <- sum(kfsllt$v[idx] ^ 2) / n
> dtf <- data.frame(r, q = qv, seta12, seta22, seps2, mll, aic, mse)
> # 表3.2
> print(dtf)
r q seta12 seta22 seps2 mll aic mse
1 2 1 0.07078040 0.000000e+00 0.1491052 -48.58750 103.1750 0.2984077
2 2 2 0.00000000 7.097290e-04 0.2432792 -54.89776 117.7955 0.3581985
3 3 2 0.08655912 5.260979e-07 0.1371066 -51.74947 113.4989 0.3234503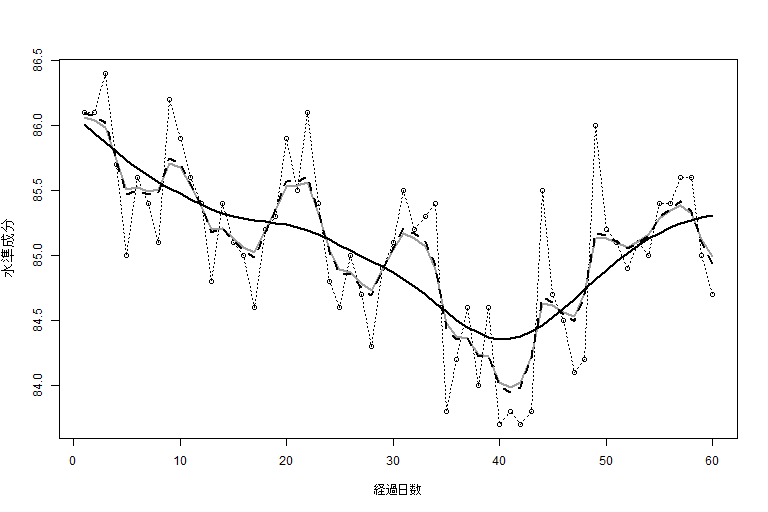
灰実線:ローカルレベルモデル, 黒実線:2次のトレンドモデル, 黒破線:ローカル線形モデル
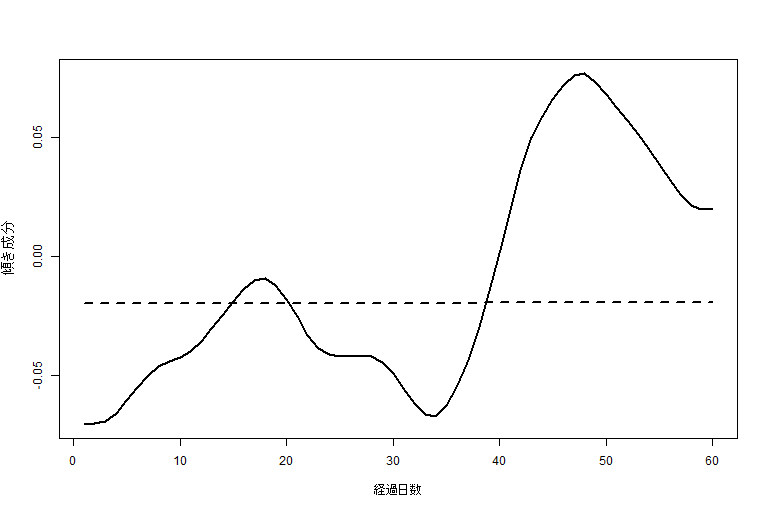
黒実線:2次のトレンドモデル, 黒破線:ローカル線形モデル